荣格工业资源APP
了解工业圈,从荣格工业资源APP开始。
传统上,人工智能在药物发现领域的应用一直受制于其对单一任务的关注,而忽视了可以丰富其预测准确性的丰富的结构化和非结构化数据。
在处理新型化合物或蛋白质时,这些局限性尤为明显,因为在这些领域中,现有知识很少或根本不存在,而人工数据注释的成本又往往高得令人望而却步……

《健康数据科学》(Health Data Science)期刊上发表的一项变革性研究介绍了一种开创性的端到端深度学习框架,即「知识驱动药物发现」(Knowledge-Empowered Drug Discovery,KEDD),旨在彻底改变药物发现领域。

药物发现旨在设计新型治疗药物,以应对某种疾病并减少其对患者的潜在副作用。对生物分子,包括药物或蛋白质的了解是药物发现过程的基础。
这些分子专业知识通常包含在三种不同的模式中:
分子结构,如分子的 SMILES 字符串和蛋白质的氨基酸序列;
来自知识图谱的结构化知识;
以及来自生物医学文档的非结构化知识。这些模式相辅相成,为制药应用研究人员提供了整体指导。
研究人员提出了用于知识驱动药物发现的端到端统一深度学习框架 KEDD,以解决上述问题。
KEDD 可同时从分子结构、知识图谱的结构化知识和生物医学文献的非结构化知识中获取生物医学专业知识;可灵活应用于各种人工智能药物发现任务。
该框架首先结合独立的现成表示学习模型,从每种模式中提取密集特征。然后,它通过串联多模态特征进行特征融合,并利用预测网络计算结果。
为了缓解结构化知识的模态缺失问题,KEDD 利用多头稀疏注意力来重建基于最相关生物分子的特征,并提出了一种模态掩蔽技术来改进稀疏注意力的训练。

图:多模态知识获取管道。(A) 稀疏注意力管道将结构特征作为查询条件,以获取 BMKG 中的前 k 个相关实体。 (B) 在 BMKG 中搜索相同的生物分子结构,以获取多模态知识。如果搜索失败或触发了模态屏蔽,就会应用稀疏注意力来重建结构化知识特征。
KEDD 侧重于药物发现所涉及的两类生物分子:药物和蛋白质。每个组成部分又包括三种模式的信息,即分子结构、结构化知识和非结构化知识。
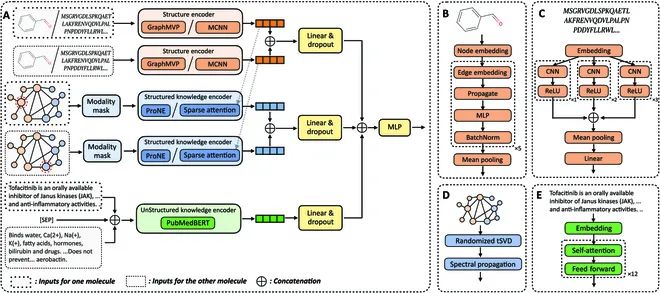
图:KEDD 架构。(A) 整体特征融合框架。根据下游任务的不同,分子的输入可以是药物、蛋白质或空。(B) 药物结构编码器 GraphMVP 的网络架构。(C) 蛋白质结构编码器 MCNN 的网络结构。(D) 结构化知识编码器 ProNE 的工作流程。(E) 非结构化知识编码器 PubMedBERT 的网络结构。
清华大学智能产业研究院的聂再清教授强调了人工智能通过 KEDD 在药物发现方面的提升潜力。这一框架将来自分子结构、知识图谱和生物医学文献的数据协同起来,提供了一种超越传统模型局限的综合方法。
KEDD 的核心是采用稳健的表征学习模型,从各种数据模式中提炼出密集的特征。其后通过融合过程整合这些特征,并利用预测网络来确定结果,从而促进其在一系列人工智能药物发现工作中的应用。
这项研究证实了 KEDD 的有效性,展示了其在关键药物发现任务中超越现有人工智能模型的能力。
展望未来,KEDD 项目的主要研究者概述了增强框架能力的宏伟计划,包括探索多模态预训练策略。该项目的总体目标是培养一个多功能、知识驱动的人工智能生态系统,加快生物医学研究,及时提供见解和建议,推动治疗发现和开发。
参考文献:
Yizhen Luo, Xing Yi Liu, Kai Yang, Kui Huang, Massimo Hong, Jiahuan Zhang, Yushuai Wu, Zaiqing Nie. Toward Unified AI Drug Discovery with Multimodal Knowledge. Health Data Sci. 2024;4:0113.DOI:10.34133/hds.0113
编译:John Xie

